极链科技分类挑战赛金牌经验分享
近期,由Kaggle主办,Leica Microsystems和NVIDIA赞助的HPAIC(Human Protein Atlas Image Classification)竞赛正式结束。比赛为期三个月,共有来自全球的2236个队伍参加,极链AI研究院与工程院最终获得挑战赛金牌。
近期,由Kaggle主办,Leica Microsystems和NVIDIA赞助的HPAIC(Human Protein Atlas Image Classification)竞赛正式结束。比赛为期三个月,共有来自全球的2236个队伍参加,极链AI研究院与工程院最终获得挑战赛金牌。
比赛介绍
蛋白质是人体细胞中的“行动者”,执行许多共同促进生命的功能。蛋白质的分类仅限于一种或几种细胞类型中的单一模式,但是为了完全理解人类细胞的复杂性,模型必须在一系列不同的人类细胞中对混合模式进行分类。
可视化细胞中蛋白质的图像通常用于生物医学研究,这些细胞可以成为下一个医学突破的关键。然而,由于高通量显微镜的进步,这些图像的生成速度远远超过人工评估的速度。因此,对于自动化生物医学图像分析以加速对人类细胞和疾病的理解,需要比以往更大的需求。
虽然这是生物学方面的竞赛,但是其本质是机器视觉方向的图像多标签分类问题,参赛队伍也包括许多机器视觉和机器学习领域的竞赛专家。
数据分析
官方给我们提供了两种类型的数据集,一部分是512x512的png图像,一部分是2048x2048或3072x3072的TIFF图像,数据集大概 268G, 其中训练集:31072 x 4张,测试集:11702 x 4张。
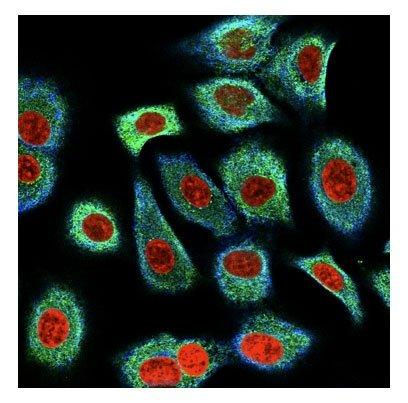
一个蛋白质图谱由4种染色方式组成(red,green,blue,yellow),图像示例如下:

我们将4个通道合并成3通道(RYB)可视化的图像如下所示:
在本次竞赛中一共有28个类别,比如 Nucleoplasm、Nuclear membrane等,每个图谱图像都可以有一个或者多个标签。标签数量统计如下:

可以发现标签数量集中在1-3个,但是仍然会有图像有5个标签,给比赛增加了一定的难度。

另一方面的难点是数据集中样本数量很不均匀,图像最多的类别有12885张,而图像最少的类别只有11张图像,这给竞赛造成很大的困难,样本数量分布情况可以在图中看出。
在比赛过程中逐步有参赛者发现官方的额外数据集HPAv18,并得到官方授权,这些数据集有105678张,很大程度的扩大了样本数量,同时给我们提供了很大的帮助。
环境资源
硬件方面我们使用了4块NVIDIA TESLA P100显卡,使用pytorch作为我们的模型训练框架。
图像预处理
HPAv18 图像与官方给出的图像有一定的差别,虽然也是由4中染色方式组成,但是每个染色图像是一个RGB图像,而不是官方的单通道图像,而且RGB三个通道的值差别较大,我们对这些图像做了预处理,对每个RGB图像只取一个通道(r_out=r,g_out=g,b_out=b,y_out=b),并将这些图像缩放到512x512和1024x1024两种尺度。

对于TIFF文件,我们用了一周的时间把这个数据集下载下来,然后将所有图像缩放到1024x1024。
数据增广
我们比赛中使用的增广方式有Rotation, Flip 和 Shear三种;因为我们不知道一张图像中的多个细胞之间是否有关联关系,所以比赛中没有使用随机裁剪的增广方式。







